In situ hybridization: Difference between revisions
(Created page with "{{Zkontrolováno | 20160302165029 | 30px | link=Uživatel:Azrael MUDr. Antonín Šípek -- WikiSkripta:Organizace|reda...") |
No edit summary |
||
| Line 1: | Line 1: | ||
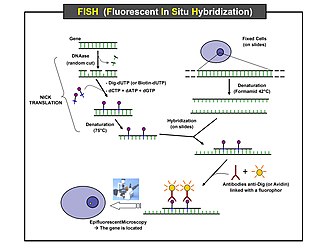
'''In situ hybridization''' belongs to molecular cytogenetic examinations. It is a method that enables the localization and identification of a specific sequence of nucleotides in DNA, or RNA. The method uses the process of DNA denaturation and reassociation. The term hybridization means that, according to the rules of complementarity, the DNA strand under investigation is connected to the second strand, which is the probe, which is usually labeled. Hybridization takes place directly in the biological material under investigation, i.e. in situ, not on isolated DNA. Chromosomes from cells in metaphase, interphase nuclei or whole cells from, for example, histological sections can be used as the examined sample. The most frequently used method of in situ hybridization is the FISH method, which uses fluorescently labeled probes. | |||
''' | |||
== | == Principle == | ||
The investigated nucleic acid must first be denatured. The hydrogen bonds between the bases are broken and the chains separate. In this way, we obtain two simple polynucleotide chains from the original DNA double helix. After induction of reassociation conditions, a short and labeled section of nucleic acid, the so-called probe, is then bound to them according to the rules of base complementarity. Binding of the probe to the DNA under investigation is manifested as a hybridization signal that can be observed under a fluorescence microscope. | |||
== | == Denaturation == | ||
[[ | Denaturation, or separation of polynucleotide chains, can be induced by the action of elevated temperature (94–96 °C), certain chemical compounds or specific enzymes. The temperature at which 50% of DNA is denatured in solution is called the '''melting point temperature (Tm)''' and depends on many factors: | ||
[[File:FISH (Fluorescent In Situ Hybridization).jpg|thumb|Scheme of the principle of the FISH (Fluorescent in situ hybridization) Experiment to localize a gene in the nucleus.]] | |||
* '''on the proportion of G–C pairs,''' | |||
** the higher it is, the higher is the Tm, | |||
* '''on the concentration of salts in the solution''' (or on the ionic strength), | |||
** the higher the concentration of salts (or monovalent cations – for example Na+), the higher the Tm value, | |||
** it is used when washing away non-specific signals (the concentration of monovalent cations decreases), | |||
* '''on the pH value of the solution,''' | |||
** significantly higher or significantly lower pH than 5-6 will destabilize DNA, | |||
* '''on the presence of denaturing agents in the solution,''' | |||
** e.g. formamide, urea, hydroxides, etc. | |||
== Method == | |||
# Material preparation (cultivation, isolation of nuclei or chromosomes, etc.) | |||
# | # Denaturation of probe and target DNA - high temperature (together or separately) | ||
# | # Hybridization of probe and target DNA - by cooling (37 °C) | ||
# | # Removal of non-specific signals (stringent washing). This is done by treating with formamide or a solution of low ionic strength while simultaneously increasing the temperature to remove the probe from sites to which it is not fully complementary. | ||
# | # Counterstaining (fluorescent staining of all chromosomes or nuclei) | ||
# Counterstaining ( | # Evaluation under a fluorescence microscope using a specialized computer program | ||
# | |||
== | == Probes == | ||
A probe is an artificially prepared, usually oligo- or polynucleotide chain that binds to the target DNA. It can be a section of DNA cloned using vectors or amplified using PCR. In order to observe the hybridization signal, the probe must be labeled | |||
=== | === Marking of the probes === | ||
* ''' | * '''Direct marking:''' | ||
** | **It allows observing the hybridization signal immediately after the end of hybridization. The disadvantage is that the signal cannot be amplified. | ||
** | **The following are most often used for direct marking: | ||
*** | *** fluorescent dye - fluorochrome (FISH method) | ||
*** | *** radioactive isotope | ||
* ''' | * '''Indirect marking:''' | ||
** | **The probe is labeled with a hapten (substance with antigenic properties), which is subsequently detected using a labeled antibody. | ||
** | **The hybridization signal can be amplified. | ||
** | **It is also possible to conjugate the antibody with an enzyme that catalyzes the formation of a color reaction. | ||
** | ** [[File:FISH locus specific probe patient.jpg|thumb|FISH locus specific probe, M. Prader-Willi, deletion 15q11-q13]]An example of a hapten is biotin, which is detected using avidin conjugated to a fluorochrome or an enzyme. | ||
=== | === Types of probes === | ||
* ''' | * '''Satellite probes''' (telomeric or centromeric) | ||
** | **They hybridize with regions containing satellite sequences. | ||
** '' | **''use'': examination of aneuploidy, identification of the origin of marker chromosomes | ||
* ''' | * '''Locus specific probe''' | ||
** | **They hybridize with unique DNA sequences (individual genes or groups of genes). | ||
** '' | **''use'': examination of microdeletions in microdeletion syndromes and malignancies and some specific translocations | ||
* ''' | * '''Painting probes''' (whole chromosome, partial) | ||
** | **They hybridize with multiple chromosomal sequences, the entire chromosome can be labeled with them. | ||
** | **Cannot be used on interphase cores. | ||
** '' | **''use'': examination of chromosomal rearrangements | ||
== Links == | |||
== | === Related articles === | ||
=== | * [[Identifikace chromozomů|Identification of chromosomes]] | ||
* [[Identifikace chromozomů]] | * [[Molekulární cytogenetika|Molecular cytogenetics]] | ||
* [[Molekulární cytogenetika]] | |||
=== | === References === | ||
* | *KOČÁREK, Eduard. ''Molekulární biologie v medicíně. ''1. vydání. Brno : Národní centrum ošetřovatelství a nelékařských zdravotnických oborů v Brně, 2007. 218 s. <nowiki>ISBN 978-80-7013-450-4</nowiki>. | ||
*KOČÁREK, Eduard a Martin PÁNEK. ''Klinická cytogenetika I : úvod do klinické cytogenetiky. ''2. vydání. Praha : Karolinum, 2010. <nowiki>ISBN 978-80-246-1880-7</nowiki>. | |||
* MAŇÁKOVÁ, Eva a Alexandra SEICHERTOVÁ. ''Metody v histologii. ''1. vydání. Praha : Karolinum, 2002. <nowiki>ISBN 80-246-0230-X</nowiki>. | |||
</ | |||
Latest revision as of 21:01, 11 January 2023
In situ hybridization belongs to molecular cytogenetic examinations. It is a method that enables the localization and identification of a specific sequence of nucleotides in DNA, or RNA. The method uses the process of DNA denaturation and reassociation. The term hybridization means that, according to the rules of complementarity, the DNA strand under investigation is connected to the second strand, which is the probe, which is usually labeled. Hybridization takes place directly in the biological material under investigation, i.e. in situ, not on isolated DNA. Chromosomes from cells in metaphase, interphase nuclei or whole cells from, for example, histological sections can be used as the examined sample. The most frequently used method of in situ hybridization is the FISH method, which uses fluorescently labeled probes.
Principle[edit | edit source]
The investigated nucleic acid must first be denatured. The hydrogen bonds between the bases are broken and the chains separate. In this way, we obtain two simple polynucleotide chains from the original DNA double helix. After induction of reassociation conditions, a short and labeled section of nucleic acid, the so-called probe, is then bound to them according to the rules of base complementarity. Binding of the probe to the DNA under investigation is manifested as a hybridization signal that can be observed under a fluorescence microscope.
Denaturation[edit | edit source]
Denaturation, or separation of polynucleotide chains, can be induced by the action of elevated temperature (94–96 °C), certain chemical compounds or specific enzymes. The temperature at which 50% of DNA is denatured in solution is called the melting point temperature (Tm) and depends on many factors:
- on the proportion of G–C pairs,
- the higher it is, the higher is the Tm,
- on the concentration of salts in the solution (or on the ionic strength),
- the higher the concentration of salts (or monovalent cations – for example Na+), the higher the Tm value,
- it is used when washing away non-specific signals (the concentration of monovalent cations decreases),
- on the pH value of the solution,
- significantly higher or significantly lower pH than 5-6 will destabilize DNA,
- on the presence of denaturing agents in the solution,
- e.g. formamide, urea, hydroxides, etc.
Method[edit | edit source]
- Material preparation (cultivation, isolation of nuclei or chromosomes, etc.)
- Denaturation of probe and target DNA - high temperature (together or separately)
- Hybridization of probe and target DNA - by cooling (37 °C)
- Removal of non-specific signals (stringent washing). This is done by treating with formamide or a solution of low ionic strength while simultaneously increasing the temperature to remove the probe from sites to which it is not fully complementary.
- Counterstaining (fluorescent staining of all chromosomes or nuclei)
- Evaluation under a fluorescence microscope using a specialized computer program
Probes[edit | edit source]
A probe is an artificially prepared, usually oligo- or polynucleotide chain that binds to the target DNA. It can be a section of DNA cloned using vectors or amplified using PCR. In order to observe the hybridization signal, the probe must be labeled
Marking of the probes[edit | edit source]
- Direct marking:
- It allows observing the hybridization signal immediately after the end of hybridization. The disadvantage is that the signal cannot be amplified.
- The following are most often used for direct marking:
- fluorescent dye - fluorochrome (FISH method)
- radioactive isotope
- Indirect marking:
- The probe is labeled with a hapten (substance with antigenic properties), which is subsequently detected using a labeled antibody.
- The hybridization signal can be amplified.
- It is also possible to conjugate the antibody with an enzyme that catalyzes the formation of a color reaction.
- An example of a hapten is biotin, which is detected using avidin conjugated to a fluorochrome or an enzyme.
Types of probes[edit | edit source]
- Satellite probes (telomeric or centromeric)
- They hybridize with regions containing satellite sequences.
- use: examination of aneuploidy, identification of the origin of marker chromosomes
- Locus specific probe
- They hybridize with unique DNA sequences (individual genes or groups of genes).
- use: examination of microdeletions in microdeletion syndromes and malignancies and some specific translocations
- Painting probes (whole chromosome, partial)
- They hybridize with multiple chromosomal sequences, the entire chromosome can be labeled with them.
- Cannot be used on interphase cores.
- use: examination of chromosomal rearrangements
Links[edit | edit source]
Related articles[edit | edit source]
References[edit | edit source]
- KOČÁREK, Eduard. Molekulární biologie v medicíně. 1. vydání. Brno : Národní centrum ošetřovatelství a nelékařských zdravotnických oborů v Brně, 2007. 218 s. ISBN 978-80-7013-450-4.
- KOČÁREK, Eduard a Martin PÁNEK. Klinická cytogenetika I : úvod do klinické cytogenetiky. 2. vydání. Praha : Karolinum, 2010. ISBN 978-80-246-1880-7.
- MAŇÁKOVÁ, Eva a Alexandra SEICHERTOVÁ. Metody v histologii. 1. vydání. Praha : Karolinum, 2002. ISBN 80-246-0230-X.